据说新出了一款叫Speedseq的基因组分析工具,可以低配快速分析人类基因组测序结果??
你好,您对这篇的文章的总结非常好。
我也在看这篇文献,对其中Methods中Excluded regions部分的原理难以理解,第一句话就不大能读懂,
Despite the high quality of the human reference
genome, artifacts remain in low-complexity regions
and unannotated paralogous s开发者_如何学Cequences that delay
processing time and confound variant interpretation.
请帮忙解惑,谢谢。
侯文俊 2021-04-15 11:59
SpeedSeq: ultra-fast personal genome analysis and interpretation
nature methods上
http://www.nature.com/nmeth/jo ... .html
开源的,可以把50X的人类基因组重测序分析在13h开发者_如何学Python内搞定,支持生殖细胞和体细胞,如果是二倍体的,会标明是纯合还是杂合。各段所需时间如下图。
这个时间是什么概念呢?一般常规的人类基因组50x重测序分析少说也要三四天(68-92小时),从下机的序列文件(fastq格式)得到最后的变异列表(通常为vcf格式,包括结构变异[SV],单碱基突变[SNP],小的插入/缺失[Indel]),甚至光比对这一步骤,就可能超过13h。如果再遇上程序崩溃等意外事件,需要人工操作恢复,则可能会更久。这太不人道了。
而且,跑分绝不是用钱堆起来的,speedseq对配置的要求并不高,一台32线程的服务器+128 GB内存就可以了。算是很亲民的成本。
至于输入输出呢,和传统的BWA-samtools-GATK-picard流程的格式并没有什么两样。
其实speedseq的比对是BWA-MEM,但把其中费时的mark duplicates步骤交给了SAMBLASTER来处理。所以会比常规的BWA要快。
在SNP和Indel方面,为了说明这个工具并非地摊货,作者们从GIAB(瓶中基因组)中找了NA12878这个人类样本来测试,发现SNP和Indel分别能达到了99.9%和89.9%的灵敏度,而误差分别为0.4%和1.1%。这个数据表现比经典的GATK-UG要好,但不如GATK-HC。
不过作者表示瓶中基因组的数据本来就是用GATK做出来的,所以GATK肯定比speedseq用的FreeBayes更有优势。于是换了一组illumina的芯片数据重新比过,speedseq果然超过GATK-HC了。
针对癌症基因组,作者找了NA12878所在的家系来测试。并和常用工具MuTect,SomaticSniper,VarScan2等横向比较。
最后发现虽然SpeedSeq的检出率不及VarScan2,但误差却在所有工具里是最低的。SpeedSeq可以检出96.4%的正义突变,以及癌症基因中98.8%的突变。
而在结构变异(SV)方面,作者采用了三种工具来互补。首先用LUMPY寻找断点,然后用自主研发的SVTyper将断点进行分类,SVTyper基于贝叶斯原理,可以注释出断点的基因型。这一点是别的工具做不到的。接下来用评价较高的CNVnator(经过优化,支持多线程)结合LUMPY和SVTyper的结果进行深度分析并对SV进行注释。效果当然也不错,看下图。
从这一系列测试可以看出,SpeedSeq的表现并不输于现有方法,但无论是资源要求还是耗时上都有极大的优势。被传统流程折磨得抓狂的筒子们不妨尝试一下。2015-08-12 3 0分享 新浪微博 qq空间 微信
最近正在测试speedseq,使用FreeBayes来得到germline mutation,确实比GATK_HC快很多。至于准确率等这段时间忙完了,在系统的比较一下。
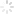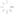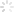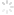 加载中,请稍侯......
加载中,请稍侯......
精彩评论